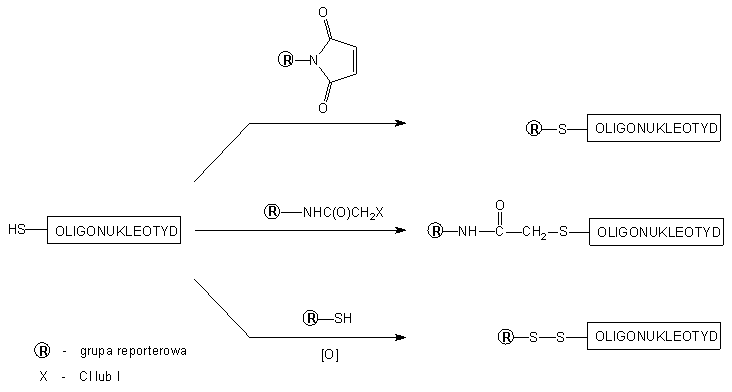Wstęp
Naturalną formą wzajemnych oddziaływań kwasów nukleinowych jest tzw. hybrydyzacja,
czyli tworzenie struktur dwu lub więcej niciowych, będących wynikiem parowania
zasad purynowych i pirymidynowych (T lub U z A, C z G). Proces ten zachodzi
z bardzo wysoką specyficznością. To ostatnie wykorzystano do wykrywania
w mieszaninach DNA lub RNA określonych sekwencji przy pomocy tzw. oligonukleotydowych
sond molekularnych. Oligonukleotydowe sondy molekularne są to oligomery
o zdefiniowanych (najczęściej programowanych) sekwencjach, które są komplementarne
do sekwencji adresowanej (poszukiwanej). W odpowiednio dobranych warunkach
sonda hybrydyzuje jedynie z sekwencją komplementarną, pomimo obecności
w badanym materiale wielu (często kilku lub kilkudziesięciu milionów) fragmentów
o wysokiej homologii do sekwencji poszukiwanej. W eksperymentach kompetycyjnych,
w których stosowano dwie krótkie sondy syntetyczne, z czego jedna była
dokładnie komplementarna do sekwencji adresowanej, a druga miała zmienioną
tylko jedną jednostkę nukleotydową, ta pierwsza tworzyła dupleks ze stukrotnie
większą wydajnością niż druga.1
Niezwykle wysoką specyficzność procesu hybrydyzacji wykorzystano w diagnostyce
genetycznej do wykrywania nawet pojedynczych mutacji w badanym materiale
(najczęściej genomowy DNA lub pula komórkowego RNA). Najprostszy protokół
hybrydyzacyjny realizowany jest w czterech etapach:
-
immobilizacja badanego materiału,
-
hybrydyzacja z sondą molekularną,
-
odmycie nadmiaru reagentów,
-
detekcja sondy.
Analiza prowadzona wg tego protokołu nie zawsze daje dostatecznie jednoznaczne
wyniki ze względu na tzw. sygnały tła. Wprowadzono więc szereg wariantowych
podejść analitycznych, których celem było podwyższenie specyficzności oddziaływań
sondy lub systemu sond z badanym DNA lub RNA2-4. Typowe przykłady
takich metod to tzw. hybrydyzacja "warstwowa" ("kanapkowa" - sandwich
hybridisation) lub jej odmiana - hybrydyzacja "prętowa" (dipstick
hybridisation) oraz hybrydyzacja "z wymianą nici" (strand displacement
hybridisation). W dwóch pierwszych przypadkach stosuje się układ dwóch
sond oligonukleotydowych, z których jedna, immobilizowana, wychwytuje fragmenty
DNA lub RNA zawierające poszukiwaną sekwencję, a drugi oligomer, sonda
detekcyjna, służy do wizualizacji utworzonego hybrydu. W metodzie "z wymianą
nici" immobilizowany układ sondy wychwytującej i tylko częściowo do niej
komplementarnej sondy detekcyjnej (chodzi o celowe utworzenie możliwie
nietrwałego dupleksu) traktuje się mieszaniną kwasów nukleinowych. Jeżeli
w analizowanym materiale obecne są sekwencje dokładnie komplementarne do
sondy wychwytującej, to te fragmenty DNA (lub RNA) wypierają sondę detekcyjną
z pierwotnego kompleksu. Porównanie ilości sondy detekcyjnej wciąż związanej
z podłożem z tą, jaka jest w roztworze, pozwala na stwierdzenie obecności
i ilościowe oszacowanie zawartości poszukiwanej sekwencji w badanym materiale.
W analityce molekularnej wykorzystuje się sondy oligonukleotydowe pochodzenia
naturalnego (kilkaset nukleotydów) lub sondy syntetyczne (zwykle o długości
10 - 50 nukleotydów). Zalety sond syntetycznych to:2,5
-
stosunkowo łatwa dostępność,
-
możliwość dowolnego programowania sekwencji sondy,
-
nie ma potrzeby denaturacji, gdyż sonda jest jednoniciowa,
-
krótki czas hybrydyzacji (minuty),
-
wysoka selektywność.
Oligonukleotydy pochodzenia naturalnego lub otrzymane na drodze chemicznej
syntezy wtedy i tylko wtedy mogą być sondami molekularnymi, kiedy można
je wykrywać. Sprawa ta jest niezwykle istotna ze względu na fakt, iż ilości
analizowanego materiału są zazwyczaj niewielkie. Popularnie stosowane bezpośrednie
metody wykrywania kwasów nukleinowych, takie jak absorpcja UV lub wybarwianie,
są dalece niewystarczające do wykrycia nawet nanomolowych ilości dupleksu
sonda-analizowany DNA lub RNA. Po to, by zwiększyć czułość detekcji, zaopatruje
się sondy molekularne w różnego typu znaczniki (atomy, grupy reporterowe),
które można wizualizować nawet przy bardzo małych stężeniach (np. attomolowych).
Dotychczas najpowszechniej stosowanymi znacznikami sond molekularnych są
radioizotopy wprowadzane do szkieletu oligonukleotydu w miejsce ich niepromieniotwórczych,
naturalnych odpowiedników. Znaczniki radioizotopowe mają tę zaletę, że
pozwalają wykrywać stosunkowo niewielkie ilości (rzędu 10-18
mola) sondy i nie zmieniają jej struktury chemicznej. Jednakże przygotowanie
i stosowanie sond znaczonych radioizotopami wiąże się z poważnymi niedogodnościami
i niebezpieczeństwami takimi jak:
-
konieczność zorganizowania laboratorium radioizotopowego,
-
konieczność odpowiedniego wyszkolenia personelu,
-
niebezpieczeństwo radioaktywnego skażenia otoczenia,
-
problem odpadów promieniotwórczych,
-
ograniczony czas życia izotopów radioaktywnych (np. t1/2 dla
32P
wynosi 14,3 dnia),
-
drogie odczynniki znakowane izotopami promieniotwórczymi,
-
możliwość radiolizy badanego materiału,
-
konieczność wielokrotnego znakowania sondy przy długich seriach badań,
-
utrudnione pomiary ilościowe.
Powyższe niedogodności sprawiły, że od kilkunastu lat trwają prace nad
alternatywnymi metodami syntezy i detekcji sond molekularnych. Działania
te są dodatkowo stymulowane potrzebami laboratoriów klinicznych, oczekujących
systemów prostych, możliwie tanich i bezpiecznych. Prace te koncentrują
się nad opracowaniem nowych metod przygotowania sond niosących znaczniki
nieradioizotopowe, które można wykrywać przez rejestrację fluorescencji,
luminescencji lub barwnej reakcji enzymatycznej.2,6-9 Pomimo
znacznej intensyfikacji badań w ostatnich latach, nie opracowano uniwersalnego
podejścia, które zyskałoby powszechną akceptację. Wciąż poszukuje się prostszych,
skuteczniejszych i tańszych systemów wprowadzania grup reporterowych do
oligonukleotydów.
Projektując nową metodę nieradioizotopowego znakowania oligonukleotydu
należy wziąć pod uwagę następujące kryteria:
-
tanie i łatwo dostępne reagenty,
-
łatwość i szybkość modyfikacji oligonukleotydu,
-
możliwość automatyzacji procesu modyfikacji,
-
trwałość sondy w warunkach hybrydyzacji,
-
wykrywalność sondy po hybrydyzacji,
-
trwałość sondy podczas przechowywania,
-
łatwość modyfikacji metody,
-
koszty.
Tabela 1
Typ grupy reporterowej
|
Znacznik
|
Wykrywalność sondy
(x 10-18 mola)
|
Metoda detekcji
|
|
Radioizotop
|
32P, 35S, 125I
|
0,05 - 6*
|
Autoradiografia,
zliczanie scyntylacyjne
|
|
Enzym
|
Peroksydaza,
alkaliczna fosfataza
|
0,1 - 10
|
Kolorymetria, fluorymetria, luminometria
|
|
Fluorofor
|
Fluoresceina, rodamina, akrydyna
|
1 - 100
|
Fluorymetria
|
|
Chelatowany jon metalu ziem rzadkich
|
Europ, terb
|
1 - 100
|
Fluorymetria
szybka
|
Jednym z podstawowych kryteriów decydujących o przydatności sondy znakowanej
nieradioizotopowo jest poziom jej detekcji, który powinien być przynajmniej
porównywalny z poziomem detekcji sondy radioizotopowej (Tabela 16,10).
Taką wykrywalność uzyskano już dla technik opartych na reakcjach enzymatycznych.11-19
Pomimo że w prostych i tanich metodach opartych na bezpośrednim pomiarze
fluorescencji, uzyskiwany sygnał jest słabszy niż dla metod radioizotopowych
czy enzymatycznych, możliwe jest jednak uzyskanie równie wysokiego poziomu
wykrywania sond fluorescencyjnych poprzez zastosowanie odpowiednich czytników
lub poprzez zwiększenie ilości badanego kwasu nukleinowego za pomocą technik
amplifikacyjnych in vitro, z których najpowszechniejszą jest łańcuchowa
reakcja polimerazy (PCR).20,21
Grupy reporterowe przyłączane są do oligomerów najczęściej za pomocą
odpowiedniego łącznika (linkera). Jest nim zwykle łańcuch polimetylenowy
zakończony grupą aktywną chemicznie (np. -NH2, -SH), wykorzystywaną
do wiązania grupy reporterowej. Wiązanie to musi być trwałe w warunkach
izolacji i dalszego stosowania sondy. W niektórych metodach przyłączanie
grup reporterowych można przeprowadzać w środowisku wodnym, co stosuje
się podczas modyfikacji fragmentów kwasów nukleinowych po usunięciu grup
ochronnych.
Grupy reporterowe typu alkilującego - imidy kwasu maleinowego i halogenoacetamidy
- przyłącza się do reszty tiolowej łącznika, wykorzystując podatność atomu
siarki na alkilację. Inną cechą reszty tiolowej, wykorzystywaną do przyłączania
grup reporterowych (np. białek), jest łatwe wytwarzanie wiązań disiarczkowych
w reakcji z drugą grupą tiolową (Rys. 1).
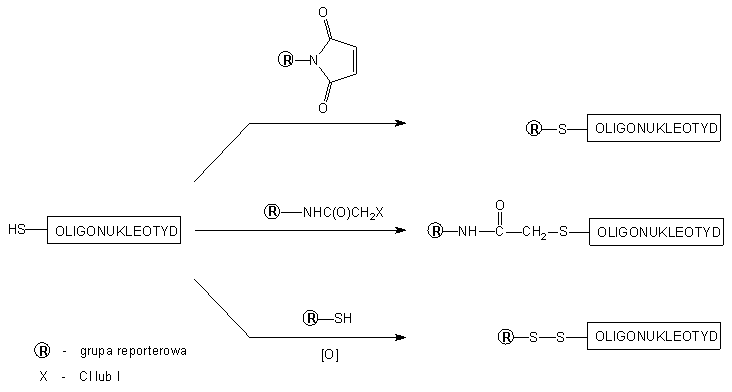
Rys. 1. Reakcje pochodnych grup reporterowych z
oligonukleotydem funkcjonalizowanym grupą tiolową
Do znakowania oligonukleotydów zmodyfikowanych ugrupowaniami z wolnymi
funkcjami aminowymi (Rys. 222) stosuje się grupy reporterowe
z ugrupowaniami acylującymi (np. izotiocyjaniany czy estry p-nitrofenylowe).
W wyniku reakcji pochodnych grup reporterowych z centrum nukleofilowym
łącznika powstaje wiązanie tioeterowe (dla reszty -SH) lub karbamidowe
czy tiomocznikowe (dla reszty -NH2). Tego typu połączenia są
trwałe w warunkach izolowania, oczyszczania i aplikacji sond molekularnych.
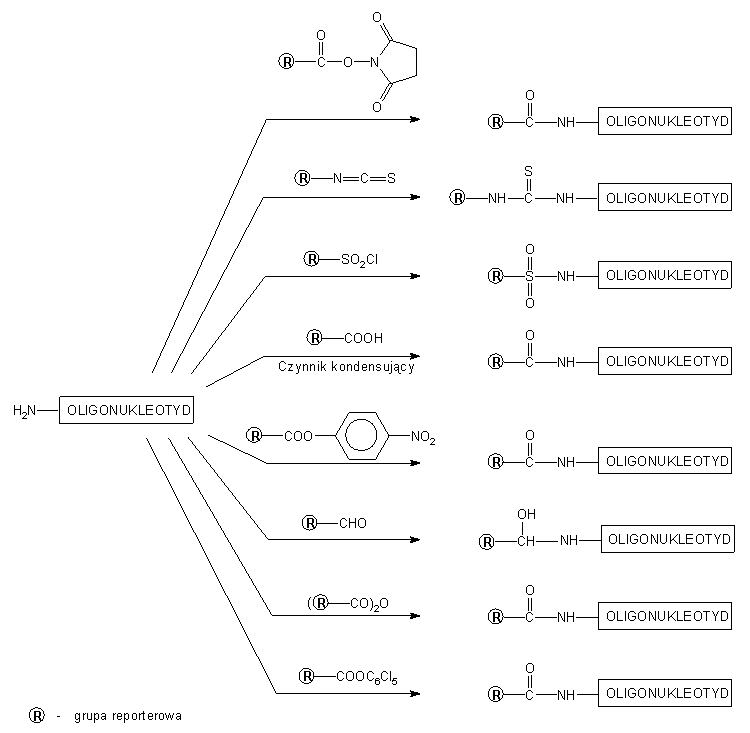
Rys. 2. Reakcje pochodnych grup reporterowych z
oligonukleotydem funkcjonalizowanym grupą aminową
Jako miejsca modyfikacji proponuje się praktycznie wszystkie elementy
struktury łańcucha oligonukleotydowego: zasady azotowe (puryny i pirymidyny),
szkielet cukrowy oraz resztę fosforanową (Rys. 3). W dalszych częściach
rozdziału omówione zostaną poszczególne podejścia i ich konsekwencje.
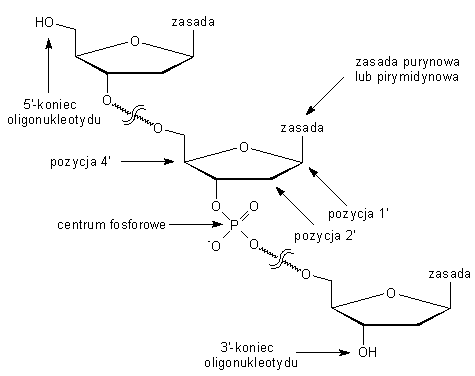
Rys. 3. Miejsca funkcjonalizacji oligonukleotydów